A caracterização da concentração de CO2 e sua variabilidade espaço-temporal é fundamental para o planejamento e execução de medidas com o propósito de mitigar as emissões de gases de efeito estufa.
O satélite OCO-2 foi lançado em órbita em julho de 2014 pela NASA, e oferece um grande potencial nas estimativas dos fluxos de dióxido de carbono (CO2). O satélite mede a concentração de CO2 atmosférico indiretamente por meio da intensidade da radiação solar refletida em função da presença de dióxido de carbono em uma coluna de ar. Desta forma, faz-se a leitura em três faixas de comprimento de onda: a do O2, na faixa de 0,757 a 0,775 μm, e as do CO2, que são subdividas em banda fraca (1,594 – 1,627 μm) e banda forte (2,043 – 2,087 μm).
Ele foi o primeiro satélite da NASA direcionado para o monitoramento dos fluxos de CO2 atmosférico, sendo um dos mais recentes, e vem apresentando usos bem diversificados, mostrando-se capaz de monitorar as emissões de combustíveis fósseis, fotossíntese, e produção de biomassa.
O objetivo desse material é apresentar os procedimentos básicos para
aquisição de dados do satélite OCO-2 e processamento inicial em R, para
tratamento, faxina, retirada de tendência e construção de mapas de
variabilidade espacial utilizando a variável concentração de dióxido de
carbono na coluna atmosférica (xco2). As análises serão realizadas
para as diferentes regiões do Brasil no período de 2014 a 2020, perído
total de dados disponibilizado pela Agencia Espacial Americana (National
Aeronautics and Space Administration - NASA).
1) Acesse o endereço https://co2.jpl.nasa.gov/
2) Acesse o Browse OCO-2 Data em Level 2 Data Set OCO-2.
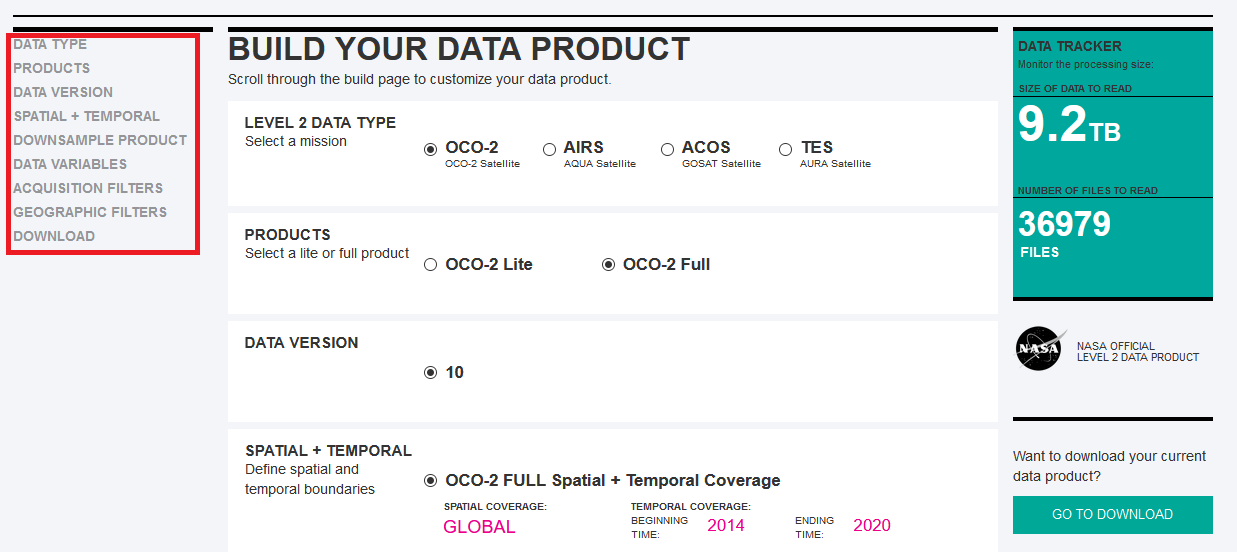
3) Role a página para baixo e acesse CUSTOMIZE PRODUCT ON BUILD PAGE.
4) No menu à esquerda estarão as 9 categorias para personalizar o banco de dados.
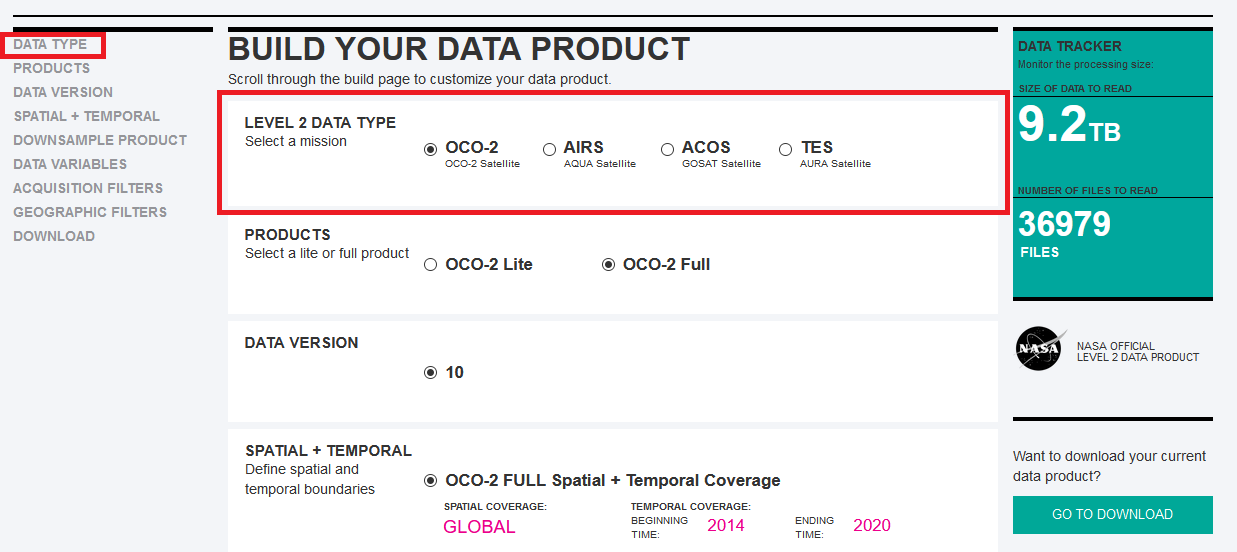
5) Em DATA TYPE selecione OCO-2 Satellite.
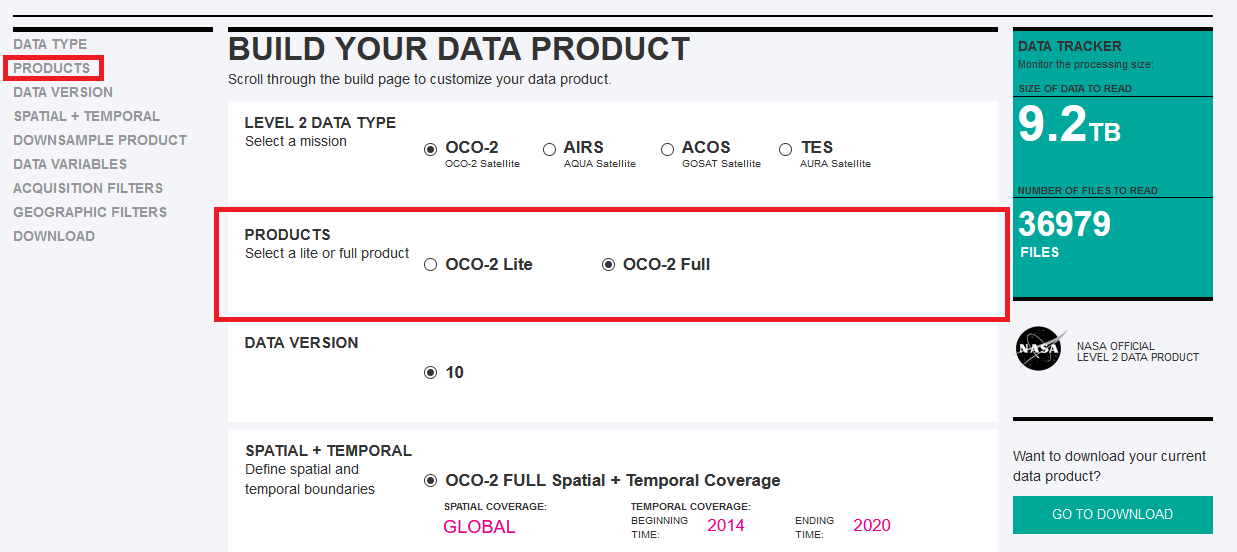
6) Em PRODUCTS selecione OCO-2 Full.
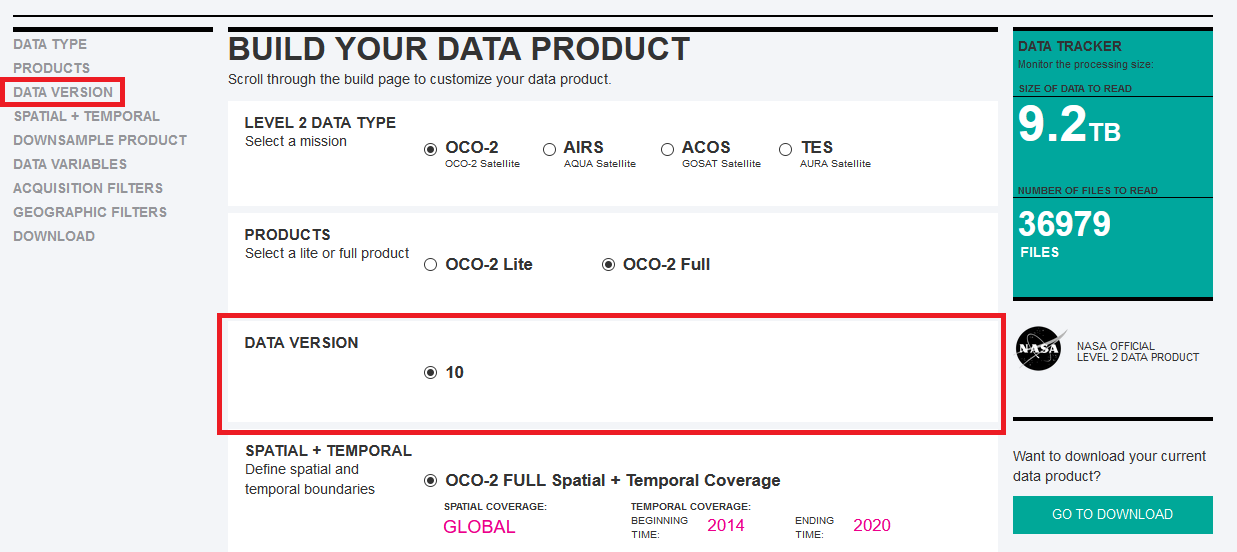
7) Em DATA VERSION selecione 10.
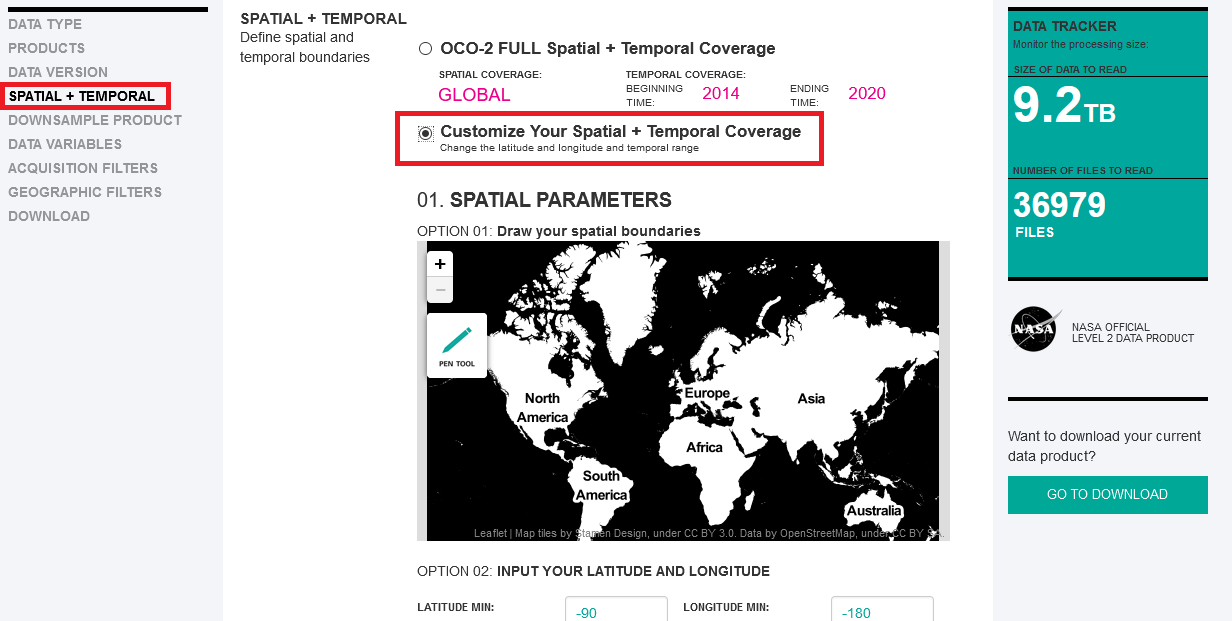
8) Em SPATIAL + TEMPORAL selecione Customize Your Spatial + Temporal Coverage.
OPTION 01 Utilize para selecionar a área para aquisição dos dados.
OPTION 02 Utilize para selecionar o período para aquisição dos dados.
9) Em DOWN SAMPLE PRODUCT selecione Yes, I want a Level 3 data product. Altere os valores das células e o passo temporal desejado.
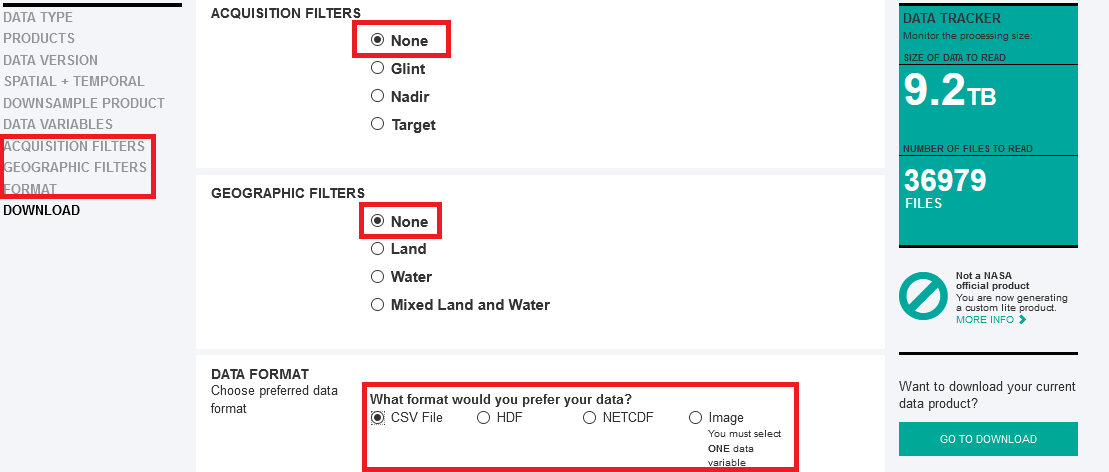
10) Em DATA VARIABLES selecione as variáveis desejadas.
11) Abaixo são apresentadas as opções para os filtros e o tipo de arquivo, selecione CSV FILE.
12) Forneça um endereço de e-mail para onde os links serão direcionados.
13) Acesse o seu e-mail, será enviado uma mensagem com o endereço dos arquivos onde você poderá acompanhar o progresso do processamento de seus dados. Ao final dessa etapa uma nova mensagem será enviada informando que os dados estão disponíveis.
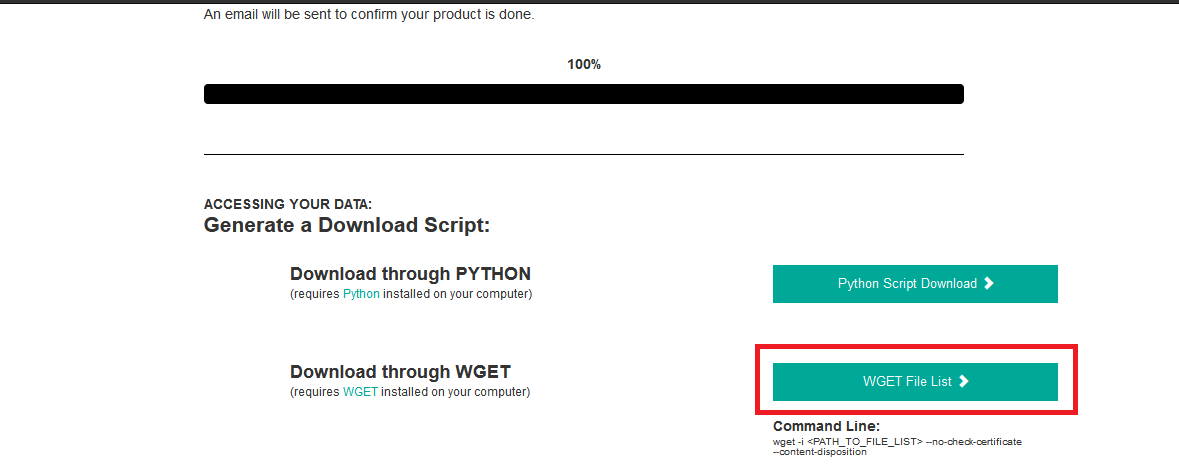
14) Acesse o link enviado em seu e-mail e você será direcionado a página.
15) Role a página para baixo e selecione a opção WGET File List.
16) Salve o arquivo .txt na pasta data-raw.
17) O arquivo contém os endereços de cada arquivo para download.
O código abaixo será utilizado para fazer o download dos arquivos CSV com os dados do satélite OCO2.
# Definindo o Pipe e funções
source("r/graficos.R")
source("r/funcoes.R")
# Links de download gerados pela NASA
links <- list.files(path = "data-raw/",
pattern = ".txt",
full.names = TRUE) |>
readr::read_table(col_names = FALSE,
col_types = "c") |>
dplyr::pull(X1)
# Definindo os caminhos e nomes dos arquivos
n_split <- lengths(stringr::str_split(links[1],"/"))
files_csv <- stringr::str_split(links,"/",simplify = TRUE)[,n_split]
files_csv <- paste0("data-raw/csv/",files_csv)
# Definindo o plano de multisession
future::plan("multisession")
# Criando a função para fazer o download dos arquivos
download_arquivos <- function(url, dir){
download.file(url, dir, mode = "wb")
return(dir)
}
tictoc::tic()
purrr::map2(links[1:1], files_csv[1:1],
purrr::possibly(download_arquivos, ""))
#> Warning in download.file(url, dir, mode = "wb"): URL
#> 'http://co2web.jpl.nasa.gov/wpsoutput/20210216_052223.726060/OCO2L2Stdv10_L3_20140905_000000_20140930_235959.csv':
#> Timeout of 60 seconds was reached
#> [[1]]
#> [1] ""
tictoc::toc()
#> 24.27 sec elapsed# Criando a função maybe_
maybe_download_nasa_prog <- function(url, dir, prog){
prog()
f <- purrr::possibly(download_arquivos, "")
f(url, dir)
}
# Rodando com a barra de progresso
progressr::with_progress({
prog <- progressr::progressor(along = links)
furrr::future_map2(links, files_csv,
maybe_download_nasa_prog, prog=prog)
})Imagem dos arquivos baixados. Observe o tamanho dos arquivo individuais, ao redor de 162 MB.
O volume de dados é alto, ao todo os arquivos somam 11 GB, então para garantir a reprodutibilidade desse material, vamos realizar uma faxina prévia dos dados, retirando os valores perdidos (falhas do sensor) que foram registrados como -999999.0.
O código abaixo realiza a faxina inicial dos dados, após sua execussão o volume de dados diminuiu consideravelmente, ao todo 63 MB, como apresentado na imagem subsequente.
faxina_co2 <- function(arquivo, col, valor_perdido){
da <- readr::read_csv(arquivo) |>
janitor::clean_names() |>
dplyr::filter({{col}} != valor_perdido)
readr::write_csv(da,arquivo)
}
purrr::map(files_csv, faxina_co2,
col=xco2_moles_mole_1,
valor_perdido= -999999)Agora vamos compilar todos os arquivos em um único que posteriormente será salvo no diretório data/oco2.rds.
oco2 <- purrr::map_dfr(files_csv, ~readr::read_csv(.x))
readr::write_rds(oco2,"data/oco2.rds")Abaixo observamos o vislumbre da base de dados, e algumas transformações serão necessárias.
dplyr::glimpse(oco2)
#> Rows: 364,529
#> Columns: 11
#> $ longitude <dbl> -74.58225, -74.58225, -74.58225…
#> $ longitude_bnds <chr> "-74.70703125:-74.4574652778", …
#> $ latitude <dbl> -30.22572489, -29.97654828, -29…
#> $ latitude_bnds <chr> "-30.3503131952:-30.1011365845"…
#> $ time_yyyymmddhhmmss <dbl> 2.014091e+13, 2.014091e+13, 2.0…
#> $ time_bnds_yyyymmddhhmmss <chr> "20140909000000:20140910000000"…
#> $ altitude_km <dbl> 3307.8, 3307.8, 3307.8, 3307.8,…
#> $ alt_bnds_km <chr> "0.0:6615.59960938", "0.0:6615.…
#> $ fluorescence_offset_relative_771nm_idp <dbl> 0.012406800, 0.010696600, -0.00…
#> $ fluorescence_offset_relative_757nm_idp <dbl> -3.58630e+00, 8.81219e-02, -3.6…
#> $ xco2_moles_mole_1 <dbl> 0.000394333, 0.000395080, 0.000…Inicialmente devemos transformar os dados de concentração de CO2,
variável xco2_moles_mole_1 para ppm em seguida devemos criar as
variáveis de data a partir da variável time_yyyymmddhhmmss.
oco2<-oco2 |>
dplyr::mutate(
xco2 = xco2_moles_mole_1*1e06,
data = lubridate::ymd_hms(time_yyyymmddhhmmss),
ano = lubridate::year(data),
mes = lubridate::month(data),
dia = lubridate::day(data),
dia_semana = lubridate::wday(data))Existe uma tendência de aumento monotônica mundial da concentração de CO2 na atmosfera, assim, ela deve ser retirada para podermos observar as tendências regionais.
oco2 |>
ggplot2::ggplot(ggplot2::aes(x=data,y=xco2)) +
ggplot2::geom_point(color="blue") +
ggplot2::geom_line(color="red")Agora devemos retirar a tendência ao longo do tempo, para isso, dentro do período específico, faremos a retirada por meio de um ajuste linear:
oco2 |>
dplyr::arrange(data) |>
dplyr::mutate(x= 1:nrow(oco2)) |>
ggplot2::ggplot(ggplot2::aes(x=x,y=xco2)) +
ggplot2::geom_point(shape=21,color="black",fill="gray") +
ggplot2::geom_smooth(method = "lm") +
ggpubr::stat_regline_equation(ggplot2::aes(
label = paste(..eq.label.., ..rr.label.., sep = "*plain(\",\")~~")))
#> Warning: The dot-dot notation (`..eq.label..`) was deprecated in ggplot2 3.4.0.
#> ℹ Please use `after_stat(eq.label)` instead.
#> This warning is displayed once every 8 hours.
#> Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
#> generated.
#> `geom_smooth()` using formula = 'y ~ x'Extrair os coeficientes a e b da análise de regressão linear (y=a+bX).
d_aux<-oco2 |>
dplyr::arrange(data) |>
dplyr::mutate(x= 1:nrow(oco2)) |>
dplyr::select(x,xco2)
mod <- lm(d_aux$xco2~d_aux$x)
summary.lm(mod)
#>
#> Call:
#> lm(formula = d_aux$xco2 ~ d_aux$x)
#>
#> Residuals:
#> Min 1Q Median 3Q Max
#> -80.677 -1.278 0.585 2.050 74.232
#>
#> Coefficients:
#> Estimate Std. Error t value Pr(>|t|)
#> (Intercept) 3.928e+02 1.216e-02 32302.8 <2e-16 ***
#> d_aux$x 4.437e-05 5.778e-08 767.8 <2e-16 ***
#> ---
#> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#>
#> Residual standard error: 3.671 on 364527 degrees of freedom
#> Multiple R-squared: 0.6179, Adjusted R-squared: 0.6179
#> F-statistic: 5.895e+05 on 1 and 364527 DF, p-value: < 2.2e-16
a<-mod$coefficients[1]
b<-mod$coefficients[2]Criando a variável xco2_est a partir da retirada da tendência.
oco2 <- oco2 |>
dplyr::arrange(data) |>
dplyr::mutate(
x= 1:nrow(oco2),
xco2_est = a + b * x,
delta = xco2_est - xco2,
XCO2 = (a-delta) - (mean(xco2) - a)
)
dplyr::glimpse(oco2)
#> Rows: 364,529
#> Columns: 21
#> $ longitude <dbl> -72.58572, -72.33615, -72.33615…
#> $ longitude_bnds <chr> "-72.7105034722:-72.4609375", "…
#> $ latitude <dbl> 6.154060, 5.157354, 5.406530, 3…
#> $ latitude_bnds <chr> "6.02947197682:6.27864858759", …
#> $ time_yyyymmddhhmmss <dbl> 2.014091e+13, 2.014091e+13, 2.0…
#> $ time_bnds_yyyymmddhhmmss <chr> "20140906000000:20140907000000"…
#> $ altitude_km <dbl> 3307.8, 3307.8, 3307.8, 3307.8,…
#> $ alt_bnds_km <chr> "0.0:6615.59960938", "0.0:6615.…
#> $ fluorescence_offset_relative_771nm_idp <dbl> 0.01034260, 0.01597090, 0.00988…
#> $ fluorescence_offset_relative_757nm_idp <dbl> 0.00158627, 0.00160726, 0.00848…
#> $ xco2_moles_mole_1 <dbl> 0.000391368, 0.000389822, 0.000…
#> $ xco2 <dbl> 391.368, 389.822, 388.482, 390.…
#> $ data <dttm> 2014-09-06 12:00:00, 2014-09-0…
#> $ ano <dbl> 2014, 2014, 2014, 2014, 2014, 2…
#> $ mes <dbl> 9, 9, 9, 9, 9, 9, 9, 9, 9, 9, 9…
#> $ dia <int> 6, 6, 6, 6, 6, 6, 6, 6, 6, 6, 6…
#> $ dia_semana <dbl> 7, 7, 7, 7, 7, 7, 7, 7, 7, 7, 7…
#> $ x <int> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, …
#> $ xco2_est <dbl> 392.8449, 392.8450, 392.8450, 3…
#> $ delta <dbl> 1.4769085, 3.0229529, 4.3629972…
#> $ XCO2 <dbl> 383.2815, 381.7354, 380.3954, 3…Visualização da concentração de CO2 no ano de 2014, primeiro ano de utilização do satélite.
oco2 |>
dplyr::filter(ano == 2014) |>
ggplot2::ggplot(ggplot2::aes(x=longitude, y=latitude, color=dia_semana)) +
ggplot2::geom_point()A próxima operação é selecionarmos na base de dados somente os pontos
pertencentes ao território brasileiro. Assim vamos utilizar o pacote
geobr para criarmos o filtro a partir do polígono do Brasil e regiões.
regiao <- geobr::read_region(showProgress = FALSE)
#> Loading required namespace: sf
#> Using year 2010
br <- geobr::read_country(showProgress = FALSE)
#> Using year 2010Agora podemos extrair os polígonos
### Polígono Brasil
pol_br <- br$geom |> purrr::pluck(1) |> as.matrix()
### Polígonos das Regiões
pol_norte <- regiao$geom |> purrr::pluck(1) |> as.matrix()
pol_nordeste <- regiao$geom |> purrr::pluck(2) |> as.matrix()
pol_sudeste <- regiao$geom |> purrr::pluck(3) |> as.matrix()
pol_sul <- regiao$geom |> purrr::pluck(4) |> as.matrix()
pol_centroeste<- regiao$geom |> purrr::pluck(5) |> as.matrix()Plot dos pontos e o polígono
br |>
ggplot2::ggplot() +
ggplot2::geom_sf(fill="#2D3E50", color="#FEBF57",
size=.15, show.legend = FALSE)+
tema_mapa() +
ggplot2::geom_point(data=oco2 |> dplyr::filter(ano == 2014) ,
ggplot2::aes(x=longitude,y=latitude),
shape=3,
col="red",
alpha=0.2)A partir da função point.in.pol() do pacote {sp}, criamos a função
abaixo para facilitar o processo de filtragem em função de um polígono
definido.
#' @title Define se pontos pertencem a um polígono
#'
#' @name def_pol
#'
#' @description Verifica para um ou mais pontos se eles se enquadram em um determinado polígono.
#'
#' @param x Vetor com a coordenada x do ponto
#' @param y Vetor com a coordenada y do ponto
#' @param pol matriz do polígono
#'
#'
#' @details Função baseada na função point.in.pol do pacote sp, utilziada
#' para filtrar os pontos pertencentes aos polígonos dos estados,
#' regiões e muicípios do Brasil.
def_pol <- function(x, y, pol){
as.logical(sp::point.in.polygon(point.x = x,
point.y = y,
pol.x = pol[,1],
pol.y = pol[,2]))
}Vamos criar o filtro para os pontos pertencentes ao polígono do Brasil e demais regiões.
oco2 <- oco2 |>
dplyr::mutate(
flag_br = def_pol(longitude, latitude, pol_br),
flag_norte = def_pol(longitude, latitude, pol_norte),
flag_nordeste = def_pol(longitude, latitude, pol_nordeste),
flag_sul = def_pol(longitude, latitude, pol_sul),
flag_sudeste = def_pol(longitude, latitude, pol_sudeste),
flag_centroeste = def_pol(longitude, latitude, pol_centroeste)
)
#> The legacy packages maptools, rgdal, and rgeos, underpinning the sp package,
#> which was just loaded, will retire in October 2023.
#> Please refer to R-spatial evolution reports for details, especially
#> https://r-spatial.org/r/2023/05/15/evolution4.html.
#> It may be desirable to make the sf package available;
#> package maintainers should consider adding sf to Suggests:.
#> The sp package is now running under evolution status 2
#> (status 2 uses the sf package in place of rgdal)
dplyr::glimpse(oco2)
#> Rows: 364,529
#> Columns: 27
#> $ longitude <dbl> -72.58572, -72.33615, -72.33615…
#> $ longitude_bnds <chr> "-72.7105034722:-72.4609375", "…
#> $ latitude <dbl> 6.154060, 5.157354, 5.406530, 3…
#> $ latitude_bnds <chr> "6.02947197682:6.27864858759", …
#> $ time_yyyymmddhhmmss <dbl> 2.014091e+13, 2.014091e+13, 2.0…
#> $ time_bnds_yyyymmddhhmmss <chr> "20140906000000:20140907000000"…
#> $ altitude_km <dbl> 3307.8, 3307.8, 3307.8, 3307.8,…
#> $ alt_bnds_km <chr> "0.0:6615.59960938", "0.0:6615.…
#> $ fluorescence_offset_relative_771nm_idp <dbl> 0.01034260, 0.01597090, 0.00988…
#> $ fluorescence_offset_relative_757nm_idp <dbl> 0.00158627, 0.00160726, 0.00848…
#> $ xco2_moles_mole_1 <dbl> 0.000391368, 0.000389822, 0.000…
#> $ xco2 <dbl> 391.368, 389.822, 388.482, 390.…
#> $ data <dttm> 2014-09-06 12:00:00, 2014-09-0…
#> $ ano <dbl> 2014, 2014, 2014, 2014, 2014, 2…
#> $ mes <dbl> 9, 9, 9, 9, 9, 9, 9, 9, 9, 9, 9…
#> $ dia <int> 6, 6, 6, 6, 6, 6, 6, 6, 6, 6, 6…
#> $ dia_semana <dbl> 7, 7, 7, 7, 7, 7, 7, 7, 7, 7, 7…
#> $ x <int> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, …
#> $ xco2_est <dbl> 392.8449, 392.8450, 392.8450, 3…
#> $ delta <dbl> 1.4769085, 3.0229529, 4.3629972…
#> $ XCO2 <dbl> 383.2815, 381.7354, 380.3954, 3…
#> $ flag_br <lgl> FALSE, FALSE, FALSE, FALSE, FAL…
#> $ flag_norte <lgl> FALSE, FALSE, FALSE, FALSE, FAL…
#> $ flag_nordeste <lgl> FALSE, FALSE, FALSE, FALSE, FAL…
#> $ flag_sul <lgl> FALSE, FALSE, FALSE, FALSE, FAL…
#> $ flag_sudeste <lgl> FALSE, FALSE, FALSE, FALSE, FAL…
#> $ flag_centroeste <lgl> FALSE, FALSE, FALSE, FALSE, FAL…Plot dos pontos do polígono pol_br.
br |>
ggplot2::ggplot() +
ggplot2::geom_sf(fill="#2D3E50", color="#FEBF57",
size=.15, show.legend = FALSE)+
tema_mapa() +
ggplot2::geom_point(data=oco2 |> dplyr::filter(flag_br, ano == 2014) ,
ggplot2::aes(x=longitude,y=latitude),
shape=3,
col="red",
alpha=0.2)Observe que houve uma falha na região nordeste, podemos então pedir os pontos presentes do polígono br ou nordeste.
br |>
ggplot2::ggplot() +
ggplot2::geom_sf(fill="#2D3E50", color="#FEBF57",
size=.15, show.legend = FALSE)+
tema_mapa() +
ggplot2::geom_point(data=oco2 |>
dplyr::filter(flag_br|flag_nordeste, ano == 2014),
ggplot2::aes(x=longitude,y=latitude),
shape=3,
col="red",
alpha=0.2)A falha continuou, então vamos corrigir o polígono da Brasil e da região nordeste.
# Retirando alguns pontos
pol_br <- pol_br[pol_br[,1]<=-34,]
pol_br <- pol_br[!((pol_br[,1]>=-38.8 & pol_br[,1]<=-38.6) &
(pol_br[,2]>= -19 & pol_br[,2]<= -16)),]
pol_nordeste <- pol_nordeste[pol_nordeste[,1]<=-34,]
pol_nordeste <- pol_nordeste[!((pol_nordeste[,1]>=-38.7 & pol_nordeste[,1]<=-38.6) & pol_nordeste[,2]<= -15),]
# Recriando o flag_nordeste
oco2 <- oco2 |>
dplyr::mutate(
flag_br = def_pol(longitude, latitude, pol_br),
flag_nordeste = def_pol(longitude, latitude, pol_nordeste)
)
# Plot do mapa e os pontos
br |>
ggplot2::ggplot() +
ggplot2::geom_sf(fill="#2D3E50", color="#FEBF57",
size=.15, show.legend = FALSE)+
tema_mapa() +
ggplot2::geom_point(data=oco2 |>
dplyr::filter(flag_br|flag_nordeste, ano == 2014),
ggplot2::aes(x=longitude,y=latitude),
shape=3,
col="red",
alpha=0.2)Agora podemos filtrar os pontos para o território brasileiro e salvar o arquivo no diretório data/oco2_br.rds.
oco2_br <- oco2 |>
dplyr::filter( flag_br | flag_nordeste ) |>
dplyr::select(-flag_br)
readr::write_rds(oco2_br,"data/oco2_br.rds")Serão as concentrações de CO2 na regição Norte onde se encontra o bioma Amazônico diferente daquele observado nas demais regiões?
oco2_br |>
tidyr::pivot_longer(
dplyr::starts_with("flag"),
names_to = "região",
values_to = "flag"
) |>
dplyr::filter(flag) |>
dplyr::mutate(região = stringr::str_remove(região,"flag_")) |>
dplyr::group_by(região, ano, mes) |>
dplyr::summarise(media_co2 = mean(XCO2, na.rm=TRUE)) |>
dplyr::mutate(
mes_ano = lubridate::make_date(ano, mes, 1)
) |>
ggplot2::ggplot(ggplot2::aes(x = mes_ano, y = media_co2,
color=região)) +
ggplot2::geom_line() +
ggplot2::theme_bw() A região
Norte apresenta uma menor sazonalidade com as menores variações da
concentração de CO2 atmosférico quando comparada às demais regiões.
A região
Norte apresenta uma menor sazonalidade com as menores variações da
concentração de CO2 atmosférico quando comparada às demais regiões.
Vamos criar a coluna região para identicação dos pontos. Além disso será criado a coluna mes_ano.
oco2_br |>
tidyr::pivot_longer(
dplyr::starts_with("flag"),
names_to = "região",
values_to = "flag"
) |>
dplyr::filter(flag) |>
dplyr::mutate(região = stringr::str_remove(região,"flag_")) |>
dplyr::group_by(região, ano, mes) |>
dplyr::summarise(media_co2 = mean(XCO2, na.rm=TRUE)) |>
dplyr::mutate(
mes_ano = lubridate::make_date(ano, mes, 1)
)
#> # A tibble: 365 × 5
#> # Groups: região, ano [35]
#> região ano mes media_co2 mes_ano
#> <chr> <dbl> <dbl> <dbl> <date>
#> 1 centroeste 2014 9 386. 2014-09-01
#> 2 centroeste 2014 10 386. 2014-10-01
#> 3 centroeste 2014 11 385. 2014-11-01
#> 4 centroeste 2014 12 382. 2014-12-01
#> 5 centroeste 2015 1 383. 2015-01-01
#> 6 centroeste 2015 2 383. 2015-02-01
#> 7 centroeste 2015 3 382. 2015-03-01
#> 8 centroeste 2015 4 382. 2015-04-01
#> 9 centroeste 2015 5 384. 2015-05-01
#> 10 centroeste 2015 6 386. 2015-06-01
#> # ℹ 355 more rowsxco2_norte <- oco2_br |>
dplyr::filter(flag_norte, ano==2014) |>
dplyr::mutate(coordxy = paste0(latitude,longitude)) |>
dplyr::group_by(longitude,latitude) |>
dplyr::summarise(xco2_media = mean(XCO2, na.rm=TRUE) )
xco2_norte |>
ggplot2::ggplot(ggplot2::aes(x=longitude, y=latitude) ) +
ggplot2::geom_point()De posse desse mapa, vamos fazer a análise de dependência espacial. Definindo os valores de X e Y e a fórmula para análise variográfica.
sp::coordinates(xco2_norte)=~ longitude+latitude
form<-xco2_media~1 # fórmula para ser utilizado na função variogramVerificando o Variograma experimental
vari_co2_norte<-gstat::variogram(form, data=xco2_norte)
vari_co2_norte |>
ggplot2::ggplot(ggplot2::aes(x=dist, y=gamma)) +
ggplot2::geom_point()Vamos ajustar um modela esférico ao variograma experimental anterior.
m_xco2 <- gstat::fit.variogram(vari_co2_norte,gstat::vgm(1,"Sph",10,0))
plot(vari_co2_norte,model=m_xco2, col=1,pl=F,pch=16)Vamos criar o grid refinado para a interpolação
x<-xco2_norte$longitude
y<-xco2_norte$latitude
dis <- 0.1 #Distância entre pontos
grid <- expand.grid(X=seq(min(x),max(x),dis), Y=seq(min(y),max(y),dis))
sp::gridded(grid) = ~ X + YKrigragem ordinária (KO)
Utilizando o algorítmo de KO, vamos estimar xco2 nos locais não amostrados.
ko_fco2<-gstat::krige(formula=form, xco2_norte, grid, model=m_xco2,
block=c(0,0),
nsim=0,
na.action=na.pass,
debug.level=-1,
)
#> [using ordinary kriging]
#> 0% done 1% done 2% done 4% done 5% done 7% done 8% done 10% done 11% done 13% done 14% done 15% done 16% done 18% done 19% done 21% done 22% done 24% done 25% done 27% done 28% done 29% done 30% done 32% done 33% done 35% done 36% done 38% done 39% done 40% done 41% done 43% done 44% done 45% done 46% done 47% done 48% done 50% done 51% done 53% done 54% done 55% done 57% done 58% done 59% done 60% done 62% done 63% done 64% done 65% done 67% done 68% done 69% done 71% done 72% done 74% done 75% done 76% done 78% done 79% done 80% done 82% done 83% done 85% done 86% done 88% done 89% done 91% done 92% done 94% done 95% done 96% done 98% done 99% done100% doneMapa de padrões espaciais para o Ano de 2014 da região Norte.
tibble::as.tibble(ko_fco2) |>
ggplot2::ggplot(ggplot2::aes(x=X, y=Y)) +
ggplot2::geom_tile(ggplot2::aes(fill = var1.pred)) +
ggplot2::scale_fill_gradient(low = "yellow", high = "blue") +
ggplot2::coord_equal()
#> Warning: `as.tibble()` was deprecated in tibble 2.0.0.
#> ℹ Please use `as_tibble()` instead.
#> ℹ The signature and semantics have changed, see `?as_tibble`.
#> This warning is displayed once every 8 hours.
#> Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
#> generated.Mapa do padrão espacial com o contorno.
tibble::as.tibble(ko_fco2) |>
dplyr::mutate(flag_norte = def_pol(X,Y,pol_norte)) |>
dplyr::filter(flag_norte) |>
ggplot2::ggplot(ggplot2::aes(x=X, y=Y),color="black") +
ggplot2::geom_tile(ggplot2::aes(fill = var1.pred)) +
ggplot2::scale_fill_gradient(low = "yellow", high = "blue") +
ggplot2::coord_equal()+
tema_mapa()+
ggplot2::labs(fill="xco2 (ppm)") +
ggspatial::annotation_scale(
location="bl",
plot_unit="km",
height = ggplot2::unit(0.2,"cm"))Criar para cada região o mapa de interpolação via krigagem ordinária por região ao longo dos trimestres dos anos de 2014 a 2020.
oco2_nest<-oco2_br |>
dplyr::mutate(trimestre = lubridate::quarter(data)) |>
tidyr::pivot_longer(
starts_with("flag"),
names_to = "região",
values_to = "flag"
) |>
dplyr::filter(flag) |>
dplyr::mutate(região = stringr::str_remove(região,"flag_")) |>
dplyr::group_by(longitude, latitude, região, ano, trimestre) |>
dplyr::summarise(media_co2 = mean(XCO2, na.rm=TRUE)) |>
dplyr::mutate(
trimestre_ano = lubridate::make_date(ano, trimestre, 1),
regi = região,
id_time = trimestre_ano
) |>
dplyr::group_by(região,trimestre_ano) |>
tidyr::nest()
head(oco2_nest)
#> # A tibble: 6 × 3
#> # Groups: região, trimestre_ano [6]
#> região trimestre_ano data
#> <chr> <date> <list>
#> 1 norte 2017-03-01 <tibble [1,476 × 7]>
#> 2 norte 2020-03-01 <tibble [1,637 × 7]>
#> 3 norte 2015-04-01 <tibble [829 × 7]>
#> 4 norte 2019-04-01 <tibble [798 × 7]>
#> 5 norte 2014-04-01 <tibble [820 × 7]>
#> 6 norte 2016-04-01 <tibble [899 × 7]>Criaremos uma função para modelagem e krigagem como apresentado anterioremente.
geoestati <- function(df,modelo="Sph"){
#Variograma
sp::coordinates(df)=~ longitude+latitude
form<-media_co2~1
vari<-gstat::variogram(form, data=df)
alpha <- max(vari$gamma)
beta <- max(vari$dist)
m_xco2 <- gstat::fit.variogram(vari,gstat::vgm(0,modelo,beta,0))
if(m_xco2$range[2] < 0 ) m_xco2 <- gstat::fit.variogram(vari,gstat::vgm(alpha,modelo,beta,0))
# Adensamento de pontos
x<-df$longitude
y<-df$latitude
dis <- 0.1 # usamos a malha padrão
grid <- expand.grid(X=seq(min(x),max(x),dis), Y=seq(min(y),max(y),dis))
sp::gridded(grid) = ~ X + Y
#Krigagem ordinária
ko_xco2<-gstat::krige(formula=form, df, grid, model=m_xco2,
block=c(0,0),
nsim=0,
na.action=na.pass
)
# Definindo os polígonos - Dependência externa da função
regiao <- df$regi[1]
if(regiao == "norte") pol_ <- pol_norte
if(regiao == "nordeste") pol_ <- pol_nordeste
if(regiao == "sul") pol_ <- pol_sul
if(regiao == "sudeste") pol_ <- pol_sudeste
if(regiao == "centroeste") pol_ <- pol_centroeste
# Saída do mapa krigado
tibble::as.tibble(ko_xco2) |>
dplyr::mutate(flag = def_pol(X,Y,pol_)) |>
dplyr::filter(flag) |>
ggplot2::ggplot(ggplot2::aes(x=X, y=Y)) +
ggplot2::geom_tile(ggplot2::aes(fill = var1.pred)) +
ggplot2::scale_fill_gradient(low = "yellow", high = "blue") +
ggplot2::coord_equal()+
tema_mapa()+
ggplot2::labs(fill="xco2 (ppm)",
title = paste(regiao,df$id_time[1]) ) +
ggspatial::annotation_scale(
location="bl",
plot_unit="km",
height = ggplot2::unit(0.2,"cm"))
}Laço para criar os mapas por trimestre.
oco2_nest <- oco2_nest |>
dplyr::mutate(
geo_sph = purrr::map(data,geoestati,modelo="Sph")
)
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
#> [using ordinary kriging]
dplyr::glimpse(oco2_nest)
#> Rows: 130
#> Columns: 4
#> Groups: região, trimestre_ano [130]
#> $ região <chr> "norte", "norte", "norte", "norte", "norte", "norte", "n…
#> $ trimestre_ano <date> 2017-03-01, 2020-03-01, 2015-04-01, 2019-04-01, 2014-04…
#> $ data <list> [<tbl_df[1476 x 7]>], [<tbl_df[1637 x 7]>], [<tbl_df[82…
#> $ geo_sph <list> [-61.83355035, -61.53355035, -61.43355035, -60.93355035…Vamos verificar alguns mapas.
print(oco2_nest$geo_sph[1])
#> [[1]]
#> Using plotunit = 'm'print(oco2_nest$geo_sph[20])
#> [[1]]
#> Using plotunit = 'm'print(oco2_nest$geo_sph[30])
#> [[1]]
#> Using plotunit = 'm'print(oco2_nest$geo_sph[60])
#> [[1]]
#> Using plotunit = 'm'print(oco2_nest$geo_sph[80])
#> [[1]]
#> Using plotunit = 'm'print(oco2_nest$geo_sph[120])
#> [[1]]
#> Using plotunit = 'm'